文摘
虽然血浆蛋白在生物过程中所扮演的重要角色,是许多药物的直接目标,遗传因素控制inter-individual血浆蛋白水平的变化不清楚。这里描述的基因架构人血浆蛋白质组在健康献血者的间隔。我们发现1927基因协会拥有1478蛋白质,现有知识增加了四倍,包括反式协会1104年的蛋白质。了解血浆蛋白水平扰动的后果,我们应用一个集成的方法,把遗传变异与生物通路,疾病,和药物数据库。我们表明,蛋白质重叠与数量性状基因表达数量性状位点,以及与疾病有关的基因座,并找到证据表明蛋白生物标志物在疾病因果角色使用孟德尔随机化分析。遗传因素与疾病通过特定的蛋白质,我们分析强调潜在的治疗靶点,机会匹配现有药物新的疾病适应症,药物在发展和潜在的安全隐患。
这是一个预览的订阅内容,通过访问你的机构
相关的文章
开放获取文章引用这篇文章。
Long-COVID患者的血浆蛋白质组表明HIF-mediated vasculo-proliferative疾病对大脑和心脏功能的影响
转化医学杂志》开放获取2023年6月10
凝固的影响因素对子宫内膜异位症的风险:孟德尔随机化研究
BMC医学开放获取2023年5月25日
细胞间粘附分子4和缺血性中风:两个示例孟德尔随机化研究
血栓形成杂志开放获取2023年4月11日
访问选项
访问其他自然组合期刊性质和54
得到自然+,请求书在线访问订阅
29.99美元/ 30天
取消任何时候
订阅本杂志
收到51印刷问题和网络访问
每年199.00美元
只有3.90美元的问题
本文租或购买
本文得到只要你需要它
39.95美元
价格可能受当地税收计算在结帐
引用
艾伯特,f . w . & Kruglyak l .监管变化复杂的角色特征和疾病。Nat。启麝猫。16,197 - 212 (2015)。
刘,y . et al . 342年人类血浆蛋白的定量变化的双胞胎人口。摩尔。系统。医学杂志。11786 (2015)。
Suhre, k . et al .连接疾病遗传风险通过人类血浆蛋白质组端点。Nat。Commun。814357 (2017)。
姚明,c . et al。全基因组关联研究血浆蛋白识别推定地因果基因,蛋白质,和心血管疾病的途径。预印在https://www.biorxiv.org/content/early/2017/05/12/136523(2017)。
德弗里斯,p . s . et al。全基因组测序研究血清肽水平:在社区研究动脉粥样硬化的风险。嗡嗡声。摩尔。麝猫。26,3442 - 3450 (2017)。
吴,l . et al .变异和基因控制蛋白质丰富的人类。自然499年,79 - 82 (2013)。
战斗,et al。监管RNA和蛋白质变化的影响。科学347年,664 - 667 (2015)。
Rohloff, j . c . et al .核酸与蛋白质像侧链配体:修改寡核苷酸适配子及其作为诊断和治疗药物。摩尔。其他。核酸3e201 (2014)。
Di Angelantonio、大肠等。不同频率的整体效率和安全性献血(间隔):45 000捐赠者的随机试验。《柳叶刀》390年,2360 - 2371 (2017)。
Astle w·j . et al。人类血液细胞性状变异的等位基因的景观和链接常见的复杂疾病。细胞167年,1415 - 1429。e19 (2016)。
伯吉斯,S。,Scott, R. A., Timpson, N. J., Davey Smith, G. & Thompson, S. G. Using published data in Mendelian randomization: a blueprint for efficient identification of causal risk factors.欧元。j .论文。30.,543 - 552 (2015)。
陌生人,b . e . et al .模式独联体监管变化多样的人群。公共科学图书馆麝猫。8e1002639 (2012)。
Lundberg, M。,Eriksson, A., Tran, B., Assarsson, E. & Fredriksson, S. Homogeneous antibody-based proximity extension assays provide sensitive and specific detection of low-abundant proteins in human blood.核酸Res。39,e102 (2011)。
沃尔特,k . et al . UK10K项目识别罕见变异在健康和疾病。自然526年,82 - 90 (2015)。
Westra周宏儒。等。系统识别反式eQTLs假定的司机已知疾病的关联。Nat麝猫。。45,1238 - 1243 (2013)。
Lappalainen, t . et al .转录组和基因组测序揭示人类功能的变化。自然501年,506 - 511 (2013)。
斯凯特,e . e . et al .映射人类肝脏基因表达的基因结构。公共科学图书馆杂志。6e107 (2008)。
西,t . et al .遗传学和超越人类单核细胞的转录组和疾病易感性。《公共科学图书馆•综合》5e10693 (2010)。
奈,a . et al . TMPRSS6 rs855791调节hepcidin体外转录和血清hepcidin水平在正常个体。血118年,4459 - 4462 (2011)。
Carrasquillo, m . m . et al .全基因组屏幕识别rs646776附近sortilin progranulin的监管机构在人血浆水平。点。j .的嗡嗡声。麝猫。87年,890 - 897 (2010)。
Gooptu B。,Dickens, J. A. & Lomas, D. A. The molecular and cellular pathology of α1抗胰蛋白酶缺乏症。地中海趋势摩尔。。20.,116 - 127 (2014)。
斯泰西,d . et al . ProGeM:候选人的框架优先顺序因果基因在分子数量性状位点。https://doi.org/10.1101/230094(2017)。
刘,j . z . et al .关联分析确定38对炎症性肠病的易感性位点,强调共享跨种群遗传风险。Nat麝猫。。47,979 - 986 (2015)。
Di Narzo a . f . et al .高通量表征IBD患者的血清蛋白质组学对衰老和遗传因素。公共科学图书馆麝猫。13e1006565 (2017)。
黄,h . et al .精细定位炎症性肠病基因座单独的变种决议。自然547年,173 - 178 (2017)。
里昂,p . a . et al .遗传学上截然不同的子集在ANCA-associated血管炎。心血管病。j .地中海。367年,214 - 223 (2012)。
默克尔,p . a . et al .识别与风险相关的功能和表达多态性anti-neutrophil细胞质autoantibody-associated血管炎。关节炎Rheumatol。69年,1054 - 1066 (2017)。
战斗中,A。,Brown, C. D., Engelhardt, B. E. & Montgomery, S. B. Genetic effects on gene expression across human tissues.自然550年,204 - 213 (2017)。
Gronberg h . et al。50 - 69岁的男性前列腺癌筛查(STHLM3):未来的以人群为基础的诊断研究。《柳叶刀》杂志。16,1667 - 1676 (2015)。
伊尔斯,r . a . et al。多个新发现的位点与前列腺癌的易感性有关。Nat麝猫。。40,316 - 321 (2008)。
主祷文,l . et al . Multi-ancestry全基因组关联研究的21000例病例和95000例对照识别新的风险位点特异反应性皮炎。Nat麝猫。。47,1449 - 1456 (2015)。
达尔,r . et al .口服MMP-9和-12抑制剂,AZD1236,生物标志物在中度/重度慢性阻塞性肺病:随机对照试验。Pulm。杂志。其他。25,169 - 177 (2012)。
甘兹,p . et al。开发和验证蛋白质对心血管风险评分结果稳定冠心病患者之一。j。医学协会。。315年,2532 - 2541 (2016)。
Traylor, m . et al .小说MMP12轨迹与大的动脉粥样硬化有关中风使用全基因组发病年龄明智的方法。公共科学图书馆麝猫。10e1004469 (2014)。
纳尔逊,m . r . et al。人类遗传的证据的支持批准药物适应症。Nat麝猫。。47,856 - 860 (2015)。
Albagha, o . m . e . et al .全基因组关联研究确定变异CSF1, OPTN和TNFRSF11A佩吉特氏病的遗传风险因素的骨头。Nat麝猫。。42,520 - 524 (2010)。
施瓦兹,P。,Rasmussen, A. Q., Kvist, T. M., Andersen, U. B. & Jørgensen, N. R. Paget’s disease of the bone after treatment with Denosumab: a case report.骨50,1023 - 1025 (2012)。
摩尔,c . et al .间隔试验确定间隔献血可以安全地和优化可以减少血液供应:随机对照试验的研究方案。试用15363 (2014)。
黄金,l . et al . Aptamer-based多路复用蛋白质组学技术对生物标志物的发现。《公共科学图书馆•综合》5e15004 (2010)。
Sattlecker, m . et al。阿尔茨海默病生物标志物发现使用SOMAscan复合蛋白技术。预防老年痴呆症。10,724 - 734 (2014)。
麦克拉伦,w . et al .推导结果的基因组变异运用API和SNP效果预测。生物信息学26,2069 - 2070 (2010)。
UniProt财团。UniProt:蛋白质信息的中心。核酸Res。43D204-D212 (2015)。
ashburn, m . et al。基因本体论财团。基因本体论:生物学的统一的工具。Nat麝猫。。2525 - 29 (2000)。
Menni, c . et al .循环蛋白质组签名的实足年龄。j . Gerontol。一个杂志。科学。医学科学。70年,809 - 816 (2015)。
非政府组织,d . et al . Aptamer-based蛋白质组学分析揭示了小说在心血管疾病的候选生物标志物和途径。循环134年,270 - 285 (2016)。
Chang, c . c . et al .第二代叮铃声:上升到更大的挑战和更丰富的数据集。Gigascience47 (2015)。
出面协调,J。豪伊,B。迈尔斯,S。,McVean, G. & Donnelly, P. A new multipoint method for genome-wide association studies by imputation of genotypes.Nat麝猫。。39,906 - 913 (2007)。
Willer c J。,Li, Y. & Abecasis, G. R. METAL: fast and efficient meta-analysis of genomewide association scans.生物信息学26,2190 - 2191 (2010)。
杨,j . et al .条件和关节multiple-SNP GWAS汇总统计分析确定额外的变异影响复杂的特征。Nat麝猫。。44,369 - 375 (2012)。
翻滚,d . et al . NHGRI GWAS目录,策划资源SNP-trait协会。核酸Res。42D1001-D1006 (2014)。
马龙,j . et al .本体建模样本变量与一个实验因素。生物信息学26,1188金宝慱官网下载112 - 1118 (2010)。
Javierre, b . m . et al . Lineage-specific基因组结构连接增强剂和非编码疾病变异基因启动子目标。细胞167年,1369 - 1384。e19 (2016)。
劳伦斯,m . et al .软件计算和注释基因组范围。公共科学图书馆第一版。医学杂志。9e1003118 (2013)。
Amberger, j·S。,Bocchini, C. A., Schiettecatte, F., Scott, A. F. & Hamosh, A. OMIM.org: Online Mendelian Inheritance in Man (OMIM®), an online catalog of human genes and genetic disorders.核酸Res。43D789-D798 (2015)。
Kanehisa, M。佐藤,Y。,Kawashima, M., Furumichi, M. & Tanabe, M. KEGG as a reference resource for gene and protein annotation.核酸Res。44D457-D462 (2016)。
d . et al . STRINGv10 Szklarczyk:蛋白质相互作用网络,集成在生命之树。核酸Res。43D447-D452 (2015)。
史密斯,r . n . et al . InterMine:一个灵活的数据仓库系统的异构生物数据的整合和分析。生物信息学28,3163 - 3165 (2012)。
a·弗兰切斯基尼et al。STRINGv9.1:蛋白质交互网络,增加覆盖率和集成。核酸Res。41D808-D815 (2013)。
列克,m . et al . 60706年分析蛋白质编码基因变异人类。自然536年,285 - 291 (2016)。
Iotchkova诉et al . GARFIELD-GWAS分析监管或功能信息的浓缩与LD修正。预印在https://www.biorxiv.org/content/early/2016/11/07/085738(2016)。
Staley j . r . et al . PhenoScanner:人类genotype-phenotype关联的数据库。生物信息学32,3207 - 3209 (2016)。
Giambartolomei, c . et al .贝叶斯检测colocalisation之间对遗传关联研究使用汇总统计。公共科学图书馆麝猫。10e1004383 (2014)。
Hingorani a &汉弗莱斯,s .大自然的随机试验。《柳叶刀》366年,1906 - 1908 (2005)。
Nikpay, m . et al . 1000年全面Genomes-based冠状动脉疾病的全基因组关联分析。Nat麝猫。。47,1121 - 1130 (2015)。
伯吉斯,S。,Butterworth, A. & Thompson, S. G. Mendelian randomization analysis with multiple genetic variants using summarized data.麝猫。论文。37,658 - 665 (2013)。
伯吉斯,S。,Dudbridge, F. & Thompson, S. G. Combining information on multiple instrumental variables in Mendelian randomization: comparison of allele score and summarized data methods.地中海统计。。35,1880 - 1906 (2016)。
伯吉斯,s & Thompson s . g .多元孟德尔随机化:使用多效性的遗传变异估计因果效应。点。j .论文。181年,251 - 260 (2015)。
伯吉斯,S。,Dudbridge, F. & Thompson, S. G. Re: “Multivariable Mendelian randomization: the use of pleiotropic genetic variants to estimate causal effects”.点。j .论文。181年,290 - 291 (2015)。
确认
a . Day-Williams j .而言,d .•迪奥戈w . Astle e Di Angelantonio e·伯尼a·理查德·j·梅森和m . Inouye评论手稿和m .锋利的帮助药物适应症GWAS特征映射。我们感谢间隔研究参与者;员工招聘NHSBT献血中心;和间隔研究协调团队,运营团队(由r·霍顿和c·摩尔)和数据管理团队(由m . Walker)。列出了资金来源的补充信息。
审核人信息
自然由于t . Lappalainen m·麦卡锡和另一个匿名的评论家(s)为他们的贡献的同行评审工作。
作者信息
作者和联系
贡献
概念化和实验设计:法学博士,A.S.B.,B.B.S.号提案,R.M.P.; methodology: B.B.S., A.S.B., J.C.M., J.E.P., H.R., S.B.; conducted experimental work: N.J., S.K.W., E.S.Z.; analysis: B.B.S., J.C.M., J.E.P., D.S., J.B., J.R.S., T.J., E.P., P.S., C.O.-W., M.A.K., S.K.W., A.C., N.B., S.L.S.; contributed reagents, materials, protocols or analysis tools: N.J., S.K.W., E.S.Z., J.B., M.A.K., J.R.S., B.P.P.; supervision: A.S.B., H.R., J.D., R.M.P., C.S.F., D.S.P., A.M.W.; writing: A.S.B., J.E.P., B.B.S., J.C.M., H.R., J.D., J.A.T., N.S., K.S.; creation of the INTERVAL BioResource: J.R.B., D.J.R.,W.H.O., N.W.M., J.D.; funding: N.W.M., J.R.B., D.J.R., W.H.O., H.R., R.M.P., J.D.; all authors critically reviewed the manuscript.
相应的作者
道德声明
相互竞争的利益
作者声明以下利益冲突:交流CSF-Merck员工;新泽西州,S.K.W., SomaLogic Inc employees and stakeholders; E.S.Z., SomaLogic Inc employee; J.C.M., R.M.P., Merck employees during this study, now Celgene employees; H.R., Merck employee during this study; J.E.P., travel and accommodation expenses and hospitality from Olink to speak at Olink-sponsored academic meetings; A.S.B., grants from Merck, Pfizer, Novartis, Biogen and Bioverativ and personal fees from Novartis; J.D., sits on the Novartis Cardiovascular and Metabolic Advisory Board, had grant support from Novartis.
额外的信息
出版商的注意:施普林格自然保持中立在发表关于司法主权地图和所属机构。
扩展数据数据和表
扩展数据图1
流程图的样品处理和质量控制阶段蛋白质组学和基因遗传分析前测量。
扩展数据图2的例子蛋白质SOMAmer非常具体的目标。
sds - page和alexa - 647标记蛋白质被IL1RL2 SOMAmer (一个)或GP1BA SOMAmer (b)。对于每个目标蛋白质,蛋白质被SOMAmer相比标准。蛋白质的同源目标是唯一可见的捕捉道,而蛋白质同源目标蛋白质显示没有证据表明绑定。这些实验被执行一次。MW标记,分子量标记。
扩展数据图3的证据蛋白质测量用SOMAscan试验的可靠性。
一个分配系数的变化在每个subcohort SOMAscan上的所有蛋白质分析。b,斯皮尔曼相关的所有蛋白质通过QC来自同时代的基线和两年的试验样品从60岁的参与者。cSOMAscan pQTL效应大小的散点图估计和Olink显示所有163 pQTLs测试(上图)和106年复制(底部)。r皮尔森相关系数。d通货膨胀因素,分布在蛋白质进行了全基因组关联测试、分层subcohort和等位基因频率(加器≥5%,加< 5%)。
扩展数据图4WFIKKN2区域是一个反式pQTL GDF11/8等离子体水平。
一个、区域关联的情节反式pQTL(前哨rs11079936变体)GDF11/8前后调整水平WFIKKN2(上部面板),和WFIKKN2独联体pQTL GDF11/8水平调整后(下半部分)。类似的模式WFIKKN2协会是见过GDF11/8调整(没有显示)。b,GDF11/8的衰减反式pQTL等离子体水平的调整独联体蛋白质WFIKKN2。
扩展数据图5 pQTLs的遗传结构。
pQTL映射n= 3301个人。一个,分布的预测结果的哨兵pQTL变体相比,匹配排列零组变异,分层独联体和反式。星号表示经验浓缩使用排列测试(10000个交换组non-associated变异)Bonferroni-corrected意义价值(P< 0.005)。栏高度代表的意思是变异的比例在每个类和误差反映的一个标准差的意思。b许多相关的蛋白质(P< 1.5×10−11与每个前哨)整个基因组变异。
扩展数据图6浓缩pQTLs DNase我高度敏感地点的组织或细胞类型。
圈显示了DNase浓缩我过敏的网站(“热点地区”)为每个55组织(183细胞类型)可以从编码和表观基因学项目,与组织或细胞类型集群和彩色解剖分组。一些组织有多个值由于多种细胞类型的可用性或多个测试每个细胞类型。辐射线显示fold-enrichment,点在圆的内边缘表示显著富集Bonferroni-corrected重要阈值P< 5×10−5。浓缩测试使用加菲尔德(测试加浓缩对变异的排列集合匹配,距离TSS和LD)。pQTL数据n= 3301个人。
扩展数据图7计划概述了合并后的“自下而上”和“自上而下”的过程用于候选基因的注释反式pQTL地区。
看到方法。GbA,牵连;KEGG京都基因和基因组的百科全书;人类,在线孟德尔遗传在人;STRINGdb,字符串的数据库。
扩展数据图8后续PR3 SOMAmers。
这些实验重复三次独立得到了类似的结果。一个与纯化PR3, SOMAmer下拉,A1AT PR3-A1AT复杂。SOMAmer PRTN3.3514.49.2丰富了PR3-A1AT复杂比自由PR3更上一层楼。相反,SOMAmer PRTN3.13720.95.3丰富自由PR3更大程度比PR3-A1AT复杂。b、解决方案的亲和力PRTN3.3514.49.2 PRTN3.13720.95.3 PR3, A1AT, PR3-A1AT复杂。SOMAmer PRTN3.3514.49.2有更高的亲和力比免费PR3 PR3-A1AT复杂。SOMAmer PRTN3.13720.95.3,另一方面,有一个免费的亲和力PR3高于SOMAmer PRTN3.3514.49.2。c、竞争结合SOMAmers PRTN3.13720.95.3 PRTN3.3514.49.2 PR3。限制数量的放射性标记PRTN3.13720.95.3孵化1 nM proteinase-3和冷PRTN3.13720.95.3或冷PRTN3.3514.49.2滴定。
扩展数据图9所示
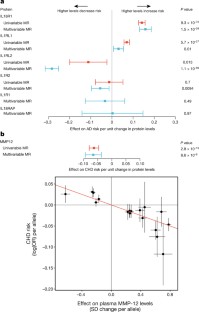
比较随机对照试验和孟德尔随机化评估之间的因果效应蛋白生物标志物水平变化对疾病风险。
扩展数据图10描述蛋白质的目标使用SOMAscan试验测量。
一个间分布与注释的蛋白质在人类蛋白质图谱进行比较。b,分子功能。
补充信息
补充信息
这个文件包含资金的细节,充分补充表传说,补充笔记和补充引用
补充图1
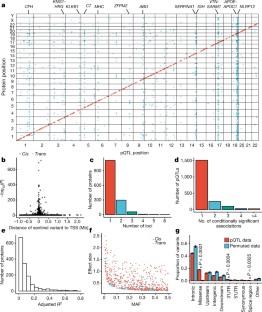
一个三维互动的哨兵variant-protein协会(red-cis blue-trans)。轴(“pQTL地位”)代表位置的哨兵变异染色体22页。轴(“蛋白质位置”)代表了起始位置的基因编码的蛋白质。z轴代表log10 (p)的关联。更多细节可以在悬停点。点击cis /反式的传奇切换显示点的cis /反式。额外的查看控件可以在窗口的右上角。为了清楚起见,对p < 10 - 300(钻石)是绘制在log10 (p) = 300。情节是使用“阴谋”生成R包v4.5.6(加拿大蒙特利尔情节Technologies Inc。)
补充表
补充表1日到21日,看到补充信息文件的完整描述
权利和权限
关于这篇文章
引用这篇文章
太阳,博博。,Maranville, J.C., Peters, J.E.et al。人血浆蛋白质组的基因组图谱。自然558年,73 - 79 (2018)。https://doi.org/10.1038/s41586 - 018 - 0175 - 2
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/s41586 - 018 - 0175 - 2
本文引用的
识别潜在的因果作用的失眠症状11409健康结果:英国生物库phenome-wide孟德尔随机分析
BMC医学(2023)
全基因组的RNA modification-related单核苷酸多态性与类风湿性关节炎有关
BMC基因组学(2023)
收敛的转录组和基因组的证据支持一个失调CXCL16和CCL5阿尔茨海默氏症
阿尔茨海默氏症的研究和治疗(2023)
凝固的影响因素对子宫内膜异位症的风险:孟德尔随机化研究
BMC医学(2023)
Long-COVID患者的血浆蛋白质组表明HIF-mediated vasculo-proliferative疾病对大脑和心脏功能的影响
转化医学杂志》(2023)
